I genomi di organismi eucariotici come il lievito sono stati usati per addestrare il modello EVO-2.Credito: Thomas Deerinck, NCMIR/Science Photo Library
Oggi gli scienziati hanno rilasciato quello che dicono è il modello di biologia per la biologia.
Il modello-che è stato addestrato su 128.000 genomi che attraversano l’albero della vita, dagli umani ai batteri a celle singole e agli archea-può scrivere cromosomi interi e piccoli genomi da zero. Può anche dare un senso al DNA esistente, comprese le varianti geniche “non codificanti” difficili da interpretare che sono legate alla malattia.
‘Chatgpt for CRISPR’ crea nuovi strumenti di modifica genica
EVO-2, co-sviluppato dai ricercatori dell’Arc Institute di Palo Alto, in California, e il produttore di chip Nvidia, è disponibile per gli scienziati attraverso interfacce Web o possono scaricare il suo codice software disponibile liberamente, i dati e altri parametri necessari per replicare il modello .
Gli sviluppatori vedono Evo-2 come una piattaforma che altri possono adattarsi ai propri usi. “Non vediamo l’ora di come scienziati e ingegneri costruiscano questo” App store “per la biologia”, ha dichiarato Patrick Hsu, bioingegnere presso l’Arc Institute e l’Università della California, Berkeley, durante un briefing stampa che annuncia il lancio di EVO-2.
Altri scienziati sono colpiti da ciò che hanno letto sul modello, che è descritto in un documento pubblicato sul sito Web dell’Arc Institute e inviato al server preprint BioRXIV. Ma dicono che dovranno calciare le gomme prima di giungere a conclusioni ferme.
“Dovremo vedere come regge i benchmark indipendenti dopo che la preprint è uscita”, afferma Anshul Kundaje, genomicista computazionale alla Stanford University di Palo Alto. Finora è colpito dall’ingegneria che è alla base del modello.
Trilioni di lettere
Negli ultimi anni, i ricercatori hanno sviluppato “modelli di linguaggio proteico” sempre più potenti come il modello ESM-3 sviluppato da ex Meta Employes che, dopo aver addestrato milioni di sequenze proteiche, sono stati usati per aiutare a prevedere le strutture proteiche e progettare totalmente nuovi Proteine tra cui redattori genici e molecole fluorescenti.

L’IA ha sognato una bufera di nuove proteine. Qualcuno di loro funziona davvero?
A differenza di questi modelli, EVO-2 è stato addestrato su dati del genoma che contiene entrambe le “sequenze di codifica”-che trasportano istruzioni per creare proteine-e DNA non codificante che include sequenze che possono controllare quando, dove e come i geni sono attivi. La prima versione di EVO pubblicata l’anno scorso è stata addestrata sui genomi di 80.000 batteri e archea – semplici organismi chiamati prokaryotes – così come i loro virus e altre sequenze.
L’ultimo modello si basa su 128.000 genomi, compresi quelli di umani e altri animali, piante e altri organismi eucariotici. Questi genomi comprendono un totale di 9,3 trilioni di lettere di DNA. Sulla base della potenza di calcolo necessaria per divorare questi dati e altre caratteristiche, EVO-2 è il più grande modello biologico di intelligenza artificiale ancora rilasciato, afferma HSU.
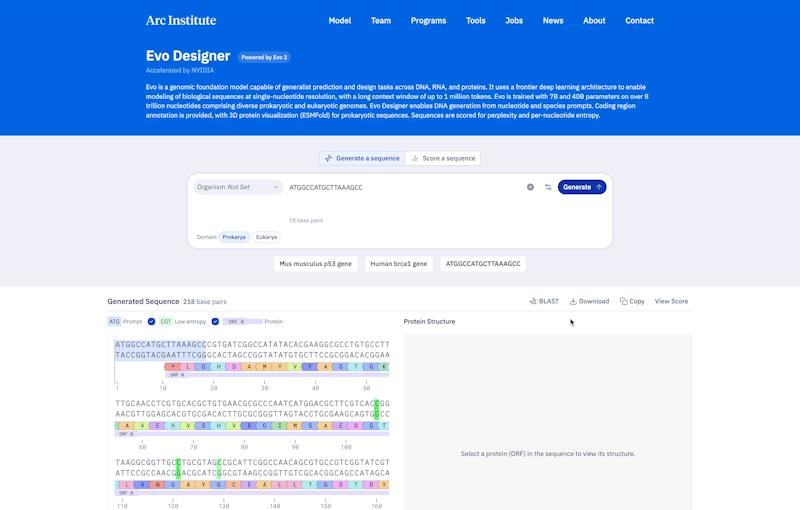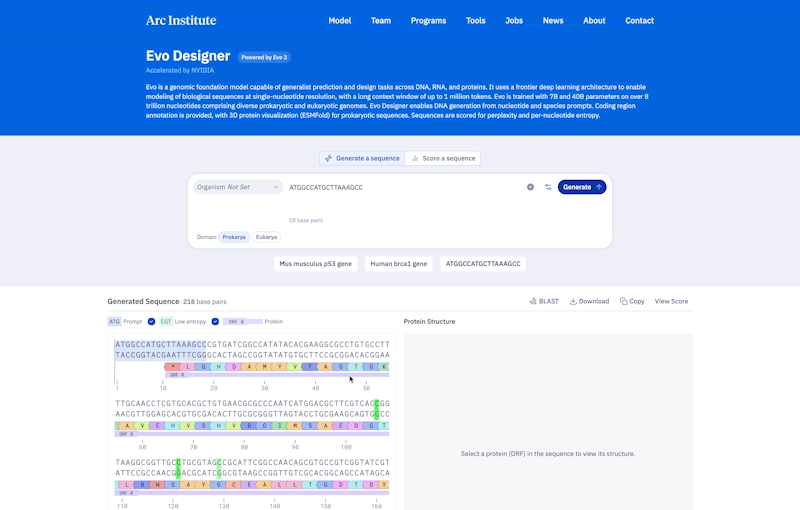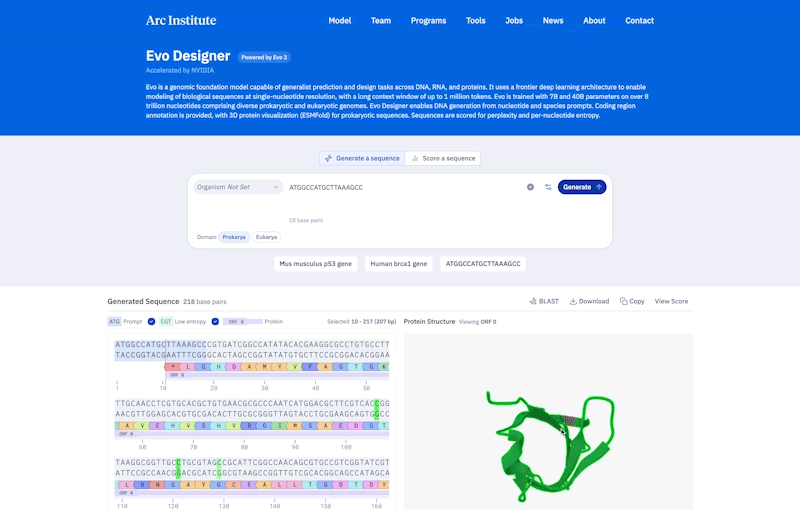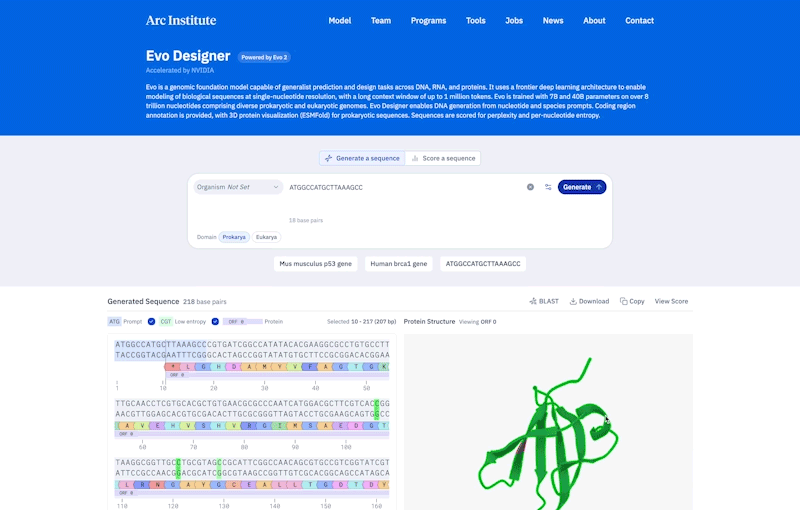
Credito: ARC Institute
Rispetto ai procarioti, i genomi eucariotici tendono ad essere più lunghi e complessi: i geni sono realizzati con segmenti intervallati di regioni di codifica e non codifica e il “DNA regolatorio” non codificante può essere lontano dai geni che controllano. Per gestire questa complessità, è stato costruito EVO-2 in modo da poter imparare modelli in sequenze di DNA fino a 1 milione di coppie di basi.
Per dimostrare la sua capacità di dare un senso a genomi complessi, HSU e i suoi colleghi hanno usato EVO-2 per prevedere gli effetti delle mutazioni precedentemente studiate in un gene implicati nel carcinoma mammario chiamato BRCA1. Ha fatto quasi i migliori modelli Bio-AI per determinare se le modifiche alle regioni di codifica avrebbero causato malattie, ha affermato HSU. “È stato dell’arte per mutazioni non codificanti.” In futuro, il modello potrebbe aiutare a identificare questi cambiamenti difficili da interpretare nei genomi dei pazienti.
I ricercatori hanno anche testato la capacità del modello di decifrare altre caratteristiche di genomi complessi, tra cui quello del mammut lanoso. “EVO-2 rappresenta un passo significativo nell’apprendimento della grammatica normativa del DNA”, afferma Christina Theodoris, una biologa computazionale dei Gladstone Institutes di San Francisco, in California.



