Quando Patrick Yizhi Cai riflette sullo stato della genomica sintetica, ricorda il grande concorso del DNA. Lanciata nel 2004, la concorrenza ha sfidato i biologi sintetici a progettare una nuova sequenza di DNA da 40.000 basi funzionale che lo sponsor del concorso, la società di sintesi del DNA US Blue Heron Biotech (ora Eurofins Genomics Blue Heron) avrebbe prodotto gratuitamente.
Non era un piccolo premio: all’epoca, producendo questa modesta lastra di DNA – meno dell’1% della lunghezza del Escherichia coli Genoma – sarebbe costato circa $ 250.000. L’obiettivo dell’azienda era quello di energizzare il campo nonscente di biologia sintetica. “Alla fine sono state ricevute domande zero”, afferma Cai, biologo sintetico presso l’Università di Manchester, nel Regno Unito. “Questo ti dice solo che anche se potessi rendere il DNA sintetico gratuitamente, nessuno aveva davvero abbastanza immaginazione 20 anni fa.”
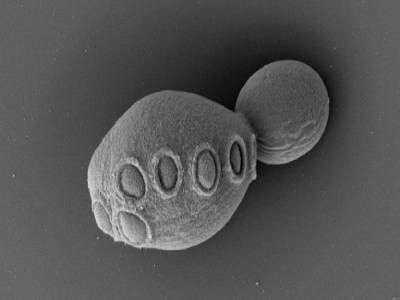
Il lievito ingegnerizzato interrompe un nuovo record: un genoma con oltre il 50% di DNA sintetico
Oggi, un progresso costante nella genomica e nella biologia computazionale-per non parlare della sintesi e dell’assemblaggio del DNA-hanno prodotto molteplici esempi di ciò che può raggiungere uno sforzo ambizioso e fantasioso per la scrittura del genoma. Il ceppo batterico sintetico JCVI-SYN3A, sviluppato al J. Craig Venter Institute (JCVI) a La Jolla, in California, è una versione aerodinamica di Mycoplasma Mycoides che sopravvive e si replica nonostante abbia avuto diverse centinaia di geni non essenziali1. Vari gruppi sono ingegneria E. coli ceppi in cui il codice genetico è stato alterato per consentire la produzione di proteine contenenti aminoacidi oltre i 20 tipicamente osservati in natura. E l’anno scorso, il Progetto multinazionale del genoma del lievito sintetico (SC2.0) ha completato la costruzione di versioni fortemente ingegnerizzate di ogni cromosoma nel lievito eucariotico in erba, Saccharomyces cerevisiae – Comprendendo circa 12 milioni di coppie di basi in tutto.
Questi sforzi sono stati preziosi esperienze di apprendimento, afferma Akos Nyerges, un ricercatore di genomica sintetica coinvolta nel E. coli Riscrivere lo sforzo nel laboratorio di George Church presso la Harvard Medical School di Boston, nel Massachusetts. “Puoi imitare e testare i passi evolutivi che altrimenti avrebbero richiesto miliardi di anni per evolversi – o non si sarebbero mai evoluti”, afferma. Ma hanno anche messo a nudo quanto non capiamo ancora sul linguaggio fondamentale del genoma. Ogni programma di rewriting del genoma finora ha affrontato sfide sostanziali e inaspettate e l’era dei genomi fatti su ordinazione rimane fuori portata. Quando si tratta di genomi fortemente modificati, afferma Nyerges, “abbiamo sottovalutato quanto sia complessa la biologia”.
Torna alle basi
La maggior parte dei progetti di genoma sintetico sono sforzi “top-down” che prendono un organismo naturale e sfogliano o ridisegnano il suo DNA. Ciò fornisce un prezioso framework iniziale relativo agli approcci “bottom-up”, in cui l’obiettivo è quello di costruire un genoma funzionante da zero. Dopotutto, spiega Farren Isaacs, un ingegnere del genoma alla Yale University di New Haven, nel Connecticut, quando si tratta di armeggiare con i genomi, il margine di errore è sorprendentemente scarso. “Se crei un errore in un gene essenziale, cancellerai l’organismo.”
Un obiettivo chiave dei progetti JCVI e SC2.0 era determinare quali geni sono veramente essenziali, una caratteristica che è sorprendentemente difficile da prevedere. John Glass, leader del programma sintetico-biologia del JCVI, afferma che quando lui e il suo team hanno pubblicato2 Il loro rapporto del 2016 sulla loro prima cellula minima, quasi un terzo dei geni rimanenti della cellula (149 su 473) non aveva una funzione nota. “Direi che ora sono 78”, aggiunge.
Per determinare quali geni erano necessari, entrambi i progetti hanno utilizzato la mutagenesi casuale – fondamentalmente, introducendo perturbazioni non mirate in tutto il genoma e chiedendo quali cellule potevano tollerare e quali gravemente hanno minato la vitalità cellulare.
Come costruire un genoma
Ma l’essenzialità è un concetto scivoloso, in particolare dato che la maggior parte dei genomi contiene licenziamenti e meccanismi di “fallimento” per ridurre al minimo l’impatto delle singole mutazioni. Il vetro e i suoi colleghi hanno incontrato dozzine di casi in cui la mutagenesi ha rivelato coppie di geni apparentemente diversi che hanno svolto inaspettatamente funzioni sovrapposte. Di conseguenza, non esiste un singolo genoma minimo, spiega. “Ne prendi uno (gene) e, con ogni scelta, stai percorrendo una strada diversa per una cella minima leggermente diversa.” Inoltre, molti geni batterici hanno più lavori, rendendo difficile riconoscere quale sia la funzione essenziale. Il vetro cita l’esempio di enolasi, un enzima con un ruolo ben noto nel metabolismo dei carboidrati che anche, a quanto pare, aiuta a degradare l’RNA indesiderato.
I “modelli computazionali” sempre più sofisticati potrebbero aiutare a rimuovere alcune delle congetture dai futuri sforzi di taglio del genoma. Nel 2020, la matematica Lucia Marucci e la biologa sintetica Claire Grierson, entrambi all’Università di Bristol, nel Regno Unito, hanno condotto uno sforzo per simulare le strategie di riduzione del genoma in un modello a cellule intere di Mycoplasma Genitalium – Un parente stretto del microrganismo a cura di JCVI3. La loro analisi, che utilizzava modelli elaborati di processi cellulari e le loro interazioni, suggeriva due riprogettazioni con set distinti di geni eliminati, ciascuno che produceva genomi che erano circa il 40% più piccoli del naturale M. genitalium genoma.
Più recentemente, Marucci e Grierson hanno iniziato a lavorare con sofisticati modelli di cellule intere di E. coli. Come descritto in una preprint del 20244i loro attuali sforzi combinano modelli meccanicistici con l’apprendimento automatico per prevedere le conseguenze della manipolazione del genoma attraverso una vasta gamma di funzioni biologiche. Questi sono descritti da migliaia di equazioni interconnesse, producendo progetti per i batteri che hanno il 40% in meno di geni rispetto ai tipi selvaggi E. coli. “Ora abbiamo un sacco di genomi ridotti minimizzati che vogliamo testare in laboratorio”, afferma Marucci.
Trova e sostituisci
Invece di fare edizioni ridotte del genoma, altri gruppi hanno deciso di riformulare sottilmente il testo genetico, incontrando una serie completamente diversa di sfide.
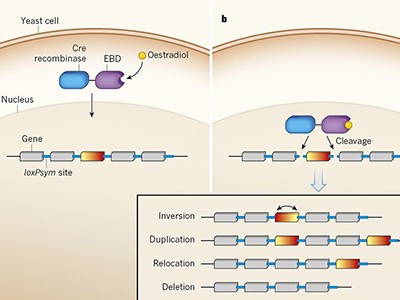
Il genoma del lievito sintetico rivela la sua versatilità
Le sequenze di codifica per proteine sono costruite in terzine nucleotidiche note come codoni. Con 61 possibili codoni per i 20 aminoacidi presenti in natura e i codoni “STOP” 3 che terminano la sintesi proteica, vi è una notevole ridondanza nel codice risultante. Varie squadre hanno dimostrato che, convertendo in modo completo ogni istanza di un determinato codone in uno dei suoi “sinonimi”, si può riutilizzare quel codone. Questo mese, ad esempio, Isaacs e i suoi colleghi hanno descritto un E. coli ceppo chiamato ocra in cui sono stati riassegnati due codoni di arresto per dirigere l’incorporazione degli aminoacidi non naturali parà-acetil-l-fenilalanina e nε-Boc-l-Lysina5. Questi aminoacidi hanno proprietà e funzioni chimiche che non esistono in natura, ma la ricodifica può anche fungere da “firewall” che impedisce l’interazione e lo scambio di materiale genetico con altri organismi in ambienti naturali.
Tale lavoro potrebbe sembrare semplice – semplicemente sostituire un codone con un altro – ma la ricodifica del genoma richiede molta pianificazione e sforzo. Dopo che i ricercatori hanno trovato tutti i casi del codone che desiderano eliminare, devono quindi capire come sostituirlo senza interrompere i geni colpiti o i macchinari regolatori. I geni batterici spesso contengono sequenze regolatorie nella sequenza di codifica delle proteine, sottolinea Nyerges e un gene su un filamento di DNA può sovrapporsi con un gene sul filo opposto. Cambiamenti apparentemente minori possono quindi avere conseguenze importanti e inaspettate.
Nyerges, Church e i loro colleghi sono alle prese con questa sfida su scala senza precedenti mentre finalizzano una variante fortemente ricodificata di E. coli che utilizza solo 57 dei 61 codoni aminoacidi naturali6. Questo sforzo ha comportato oltre 73.000 modifiche al genoma a 4 megabase del ceppo, che inevitabilmente crea effetti non intenzionali. “Alcune cose accadranno prontamente senza alcun impatto sulla crescita o sulla forma fisica, mentre altre hanno un impatto sorprendente”, afferma Nyerges. Alcune modifiche disabilitate gli elementi normativi esistenti o ne sono stati creati inconsapevolmente; Altri hanno stabilito nuove sequenze di codifica per proteine. “E stiamo solo imparando questo mentre andiamo.”
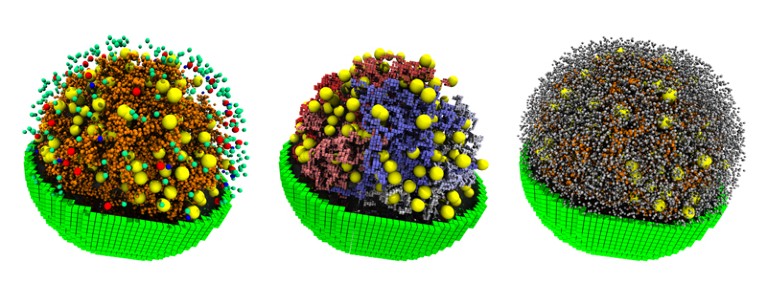
Modelli generati da computer della cellula minima sintetica creata dai ricercatori del J. Craig Venter Institute di La Jolla, in California.Credito: laboratorio di Zaida Luthey-Schulten
Sordinare questi problemi è un’impresa sostanziale a sé stante. Ad esempio, durante il processo di ricodifica per la loro tensione ocra, Isaacs e il suo team hanno utilizzato ampie analisi “multi-omiche” per caratterizzare il batterio. “Abbiamo raccolto dati di profilazione metabolica in diverse condizioni (cultura)”, afferma. “Abbiamo anche raccolto dati di proteomica confrontando la cellula ricodificata con alcuni progenitori diversi, comprese le cellule di tipo selvaggio.” In questo modo, hanno sistematicamente modificato il genoma fino a quando le cellule non sono state in grado di crescere in condizioni di coltura standard all’incirca allo stesso ritmo dei batteri non modificati-un risultato non banale, dato che il genoma che ricodifica spesso compromette la crescita. Anche Nyerges e i suoi colleghi si sono rivolti a Multi-Omics per risolvere il loro genoma a 57 codon. Hanno anche usato una strategia sperimentale che stimola la rapida evoluzione dei batteri in cultura, per promuovere la selezione di mutazioni del genoma che migliorano la forma fisica.
Gli strumenti algoritmici aiutano anche i ricercatori a modellare e prevedere in anticipo i risultati di alcuni esperimenti di rewriting del genoma. Ad esempio, il team di biologo sintetico Howard Salis presso la Pennsylvania State University di University Park utilizza dati quantitativi da schermi ad alto rendimento di cellule geneticamente modificate e fili di DNA sintetico per sviluppare algoritmi che possono definire, caratterizzare, caratterizzare sequenze che governano i processi come come trascrizione e traduzione. “Un documento tipico per noi al giorno d’oggi è ovunque da 10.000 a 100.000 esperimenti progettati e definiti diversi”, afferma Salis. I risultati vengono utilizzati per estrarre principi fisici veribili che consentono agli algoritmi di prevedere, ad esempio, come le modifiche alla sequenza del promotore di un gene alterano l’espressione a valle.
“Puoi fare a terra tutto”, afferma Salis. “E possiamo combinare i nostri modelli esistenti per progettare i prossimi esperimenti per comprendere le rimanenti cose incomprese.” In effetti, il laboratorio della chiesa ha usato alcuni strumenti di Salis per progettare il suo microbo a 57 codon. Nyerges afferma che tali algoritmi sono stati una risorsa sostanziale, sebbene non sufficiente per prevenire una notevole risoluzione dei problemi. “Anche cambiamenti molto minuscoli possono portare a cumulativamente a significativi problemi di fitness una volta che si sommano migliaia di geni in un genoma”, afferma.



